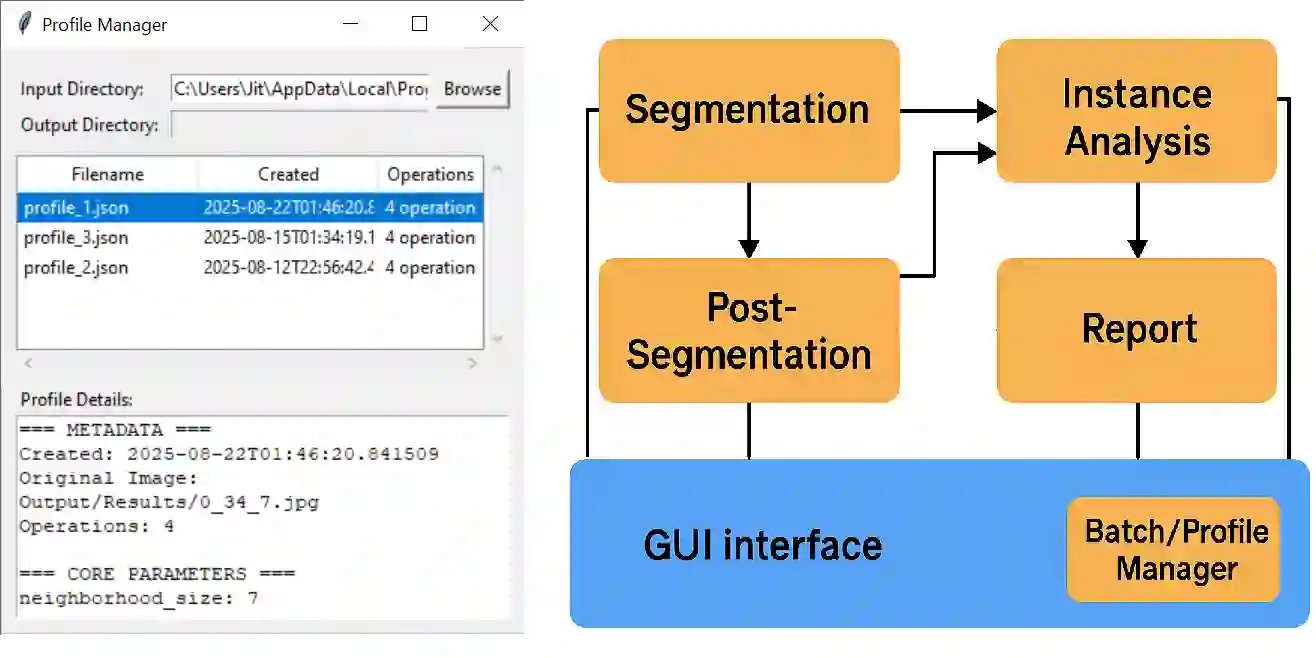
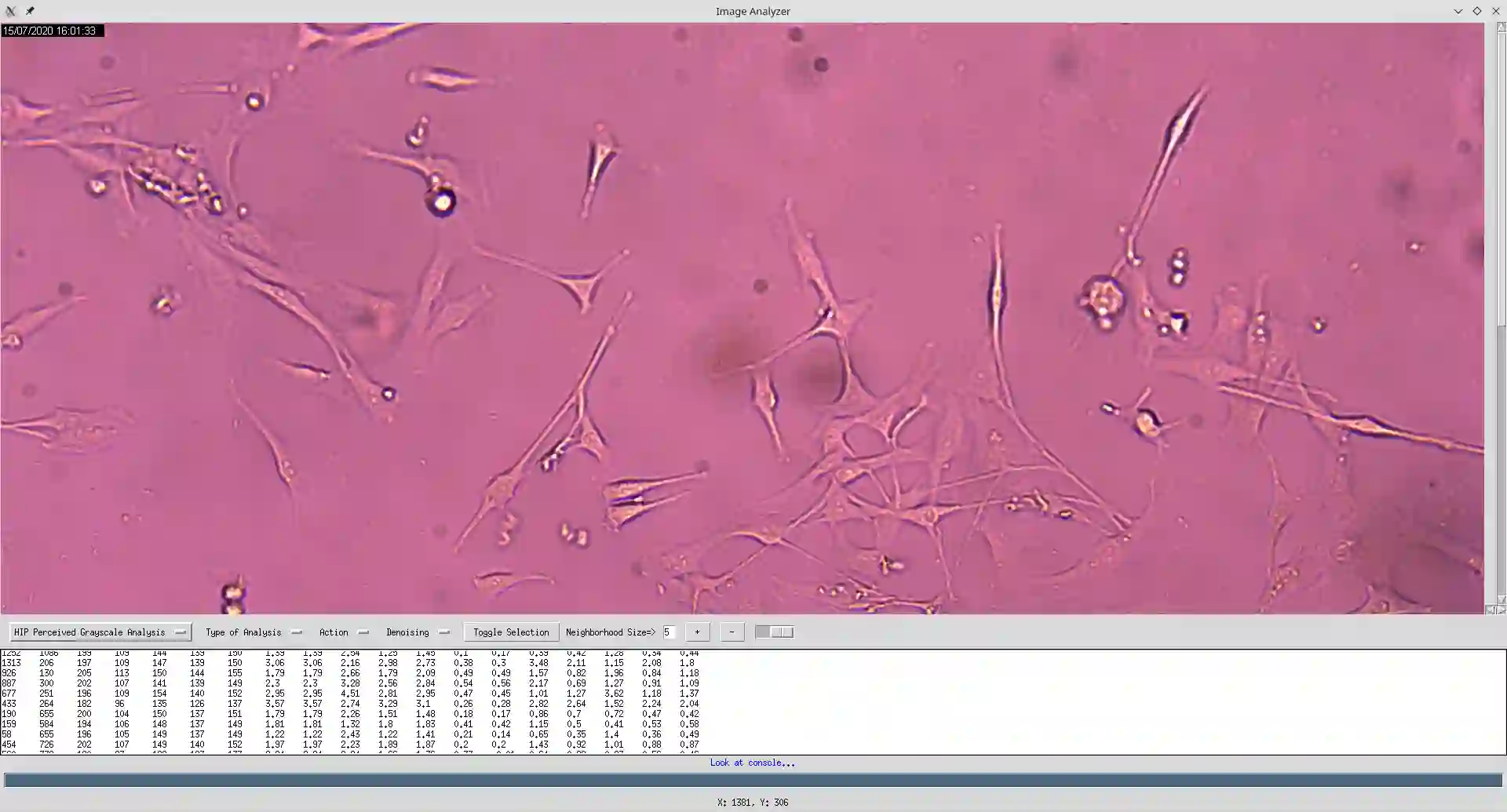
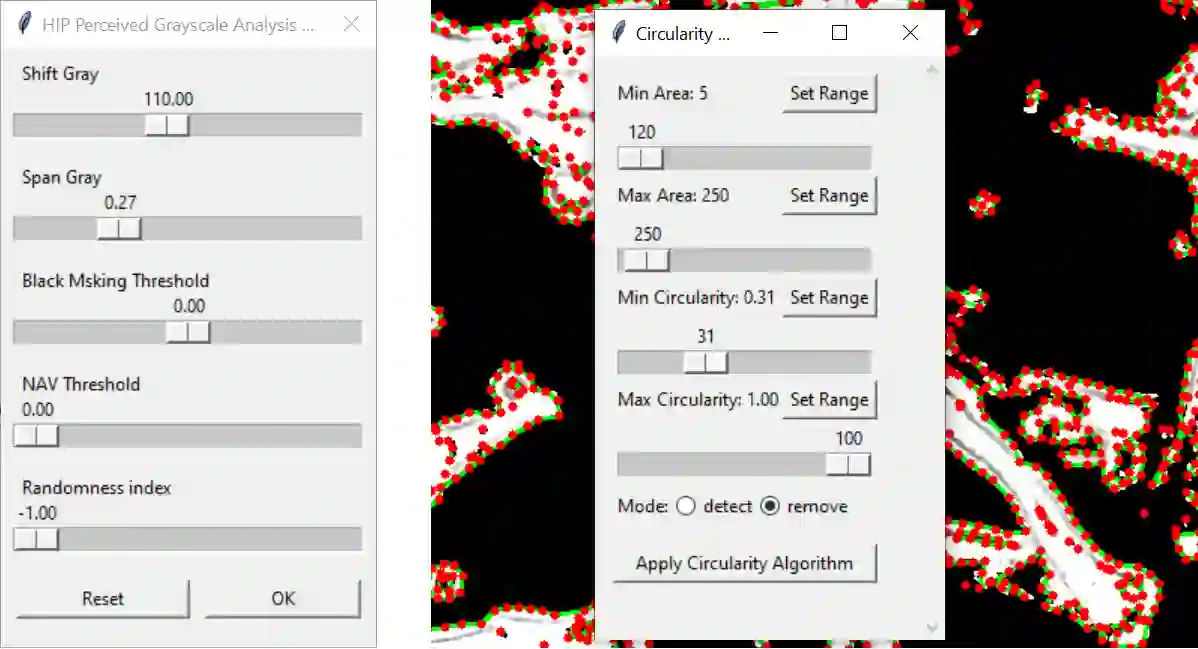
Bright-field microscopy, a cost-effective solution for live-cell culture, is often the only resource available, along with standard CPUs, for many low-budget labs. The inherent challenges of bright-field images -- their noisiness, low contrast, and dynamic morphology -- coupled with a lack of GPU resources and complex software interfaces, hinder the desired research output. This article presents a novel microscopy image analysis framework designed for low-budget labs equipped with a standard CPU desktop. The Python-based program enables cytometric analysis of live, unstained cells in culture through an advanced computer vision and machine learning pipeline. Crucially, the framework operates on label-free data, requiring no manually annotated training data or training phase. It is accessible via a user-friendly, cross-platform GUI that requires no programming skills, while also providing a scripting interface for programmatic control and integration by developers. The end-to-end workflow performs semantic and instance segmentation, feature extraction, analysis, evaluation, and automated report generation. Its modular architecture supports easy maintenance and flexible integration while supporting both single-image and batch processing. Validated on several unstained cell types from the public dataset of livecells, the framework demonstrates superior accuracy and reproducibility compared to contemporary tools like Cellpose and StarDist. Its competitive segmentation speed on a CPU-based platform highlights its significant potential for basic research and clinical applications -- particularly in cell transplantation for personalised medicine and muscle regeneration therapies. The access to the application is available for reproducibility
翻译:明场显微成像作为一种经济高效的活细胞培养观测手段,常与标准CPU计算设备共同构成许多低预算实验室的唯一可用资源。明场图像固有的噪声干扰、低对比度及动态形态学特征,加之GPU资源匮乏与复杂软件界面的限制,严重制约了相关研究的预期产出。本文提出一种专为配备标准CPU台式机的低预算实验室设计的显微图像分析框架。该基于Python的程序通过先进的计算机视觉与机器学习流程,实现对培养环境中未染色活细胞的细胞计量分析。该框架的核心优势在于其无需标记数据即可运行,既不依赖人工标注的训练数据,也无需训练阶段。系统提供无需编程技能即可操作的跨平台友好图形界面,同时为开发者保留了脚本接口以实现程序化控制与集成。其端到端工作流涵盖语义与实例分割、特征提取、分析评估及自动化报告生成。模块化架构支持简易维护与灵活集成,并兼容单张图像与批量处理模式。通过在公开活细胞数据集(livecells)中多种未染色细胞类型上的验证,本框架相较于Cellpose、StarDist等当代工具展现出更优的准确性与可重复性。其在CPU平台上的竞争优势分割速度,彰显了该系统在基础研究与临床应用领域的巨大潜力——尤其在个性化医疗的细胞移植与肌肉再生治疗方向。该应用程序已开放获取以确保可重复性验证。