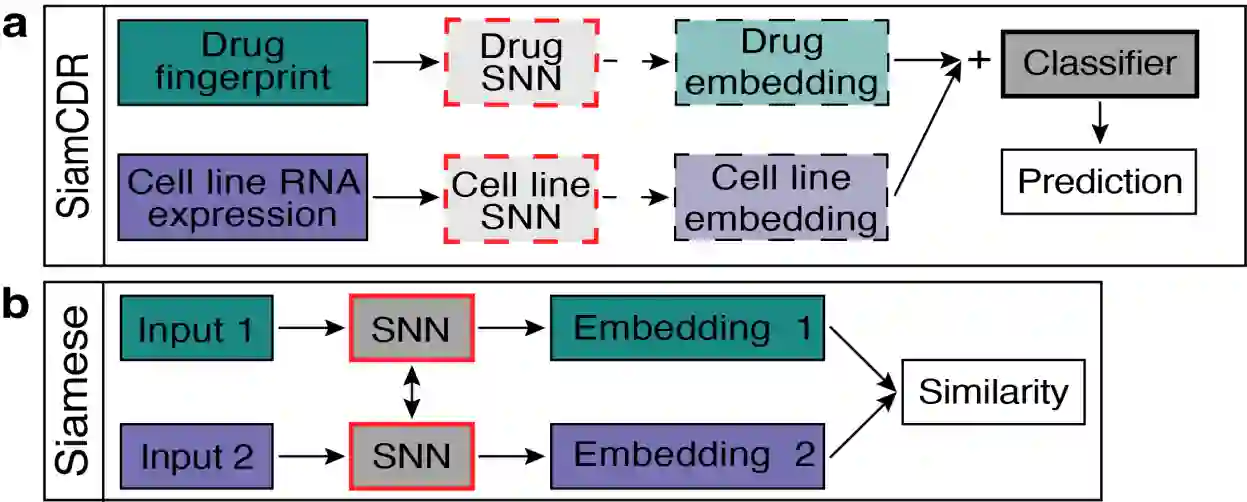
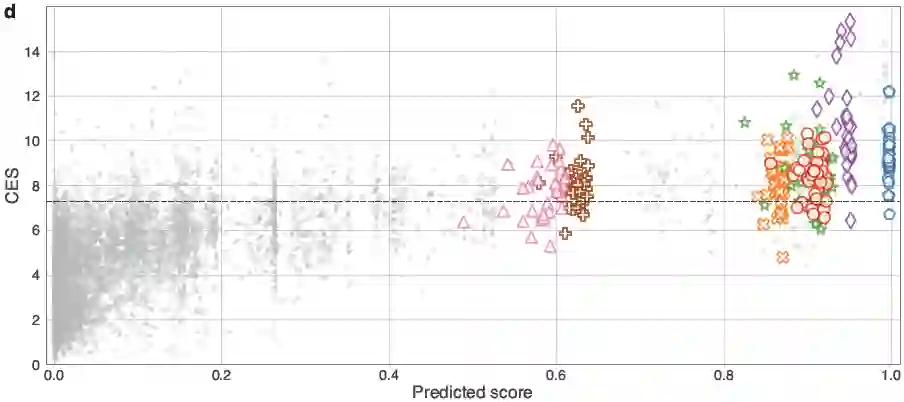
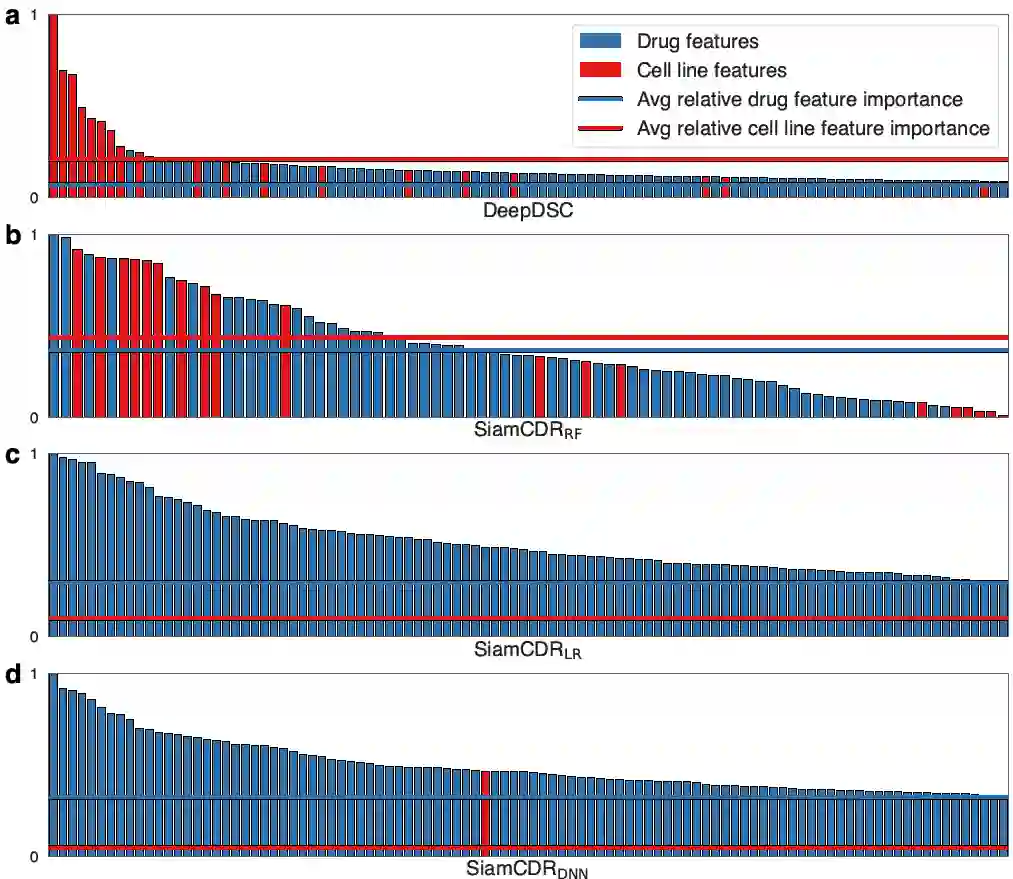
Due to cancer's complex nature and variable response to therapy, precision oncology informed by omics sequence analysis has become the current standard of care. However, the amount of data produced for each patients makes it difficult to quickly identify the best treatment regimen. Moreover, limited data availability has hindered computational methods' abilities to learn patterns associated with effective drug-cell line pairs. In this work, we propose the use of contrastive learning to improve learned drug and cell line representations by preserving relationship structures associated with drug mechanism of action and cell line cancer types. In addition to achieving enhanced performance relative to a state-of-the-art method, we find that classifiers using our learned representations exhibit a more balances reliance on drug- and cell line-derived features when making predictions. This facilitates more personalized drug prioritizations that are informed by signals related to drug resistance.
翻译:暂无翻译